Выбор признаков с помощью генетических алгоритмов (пример)
Материал из MachineLearning.
(→Исходный код) |
(→Исходный код) |
||
| Строка 95: | Строка 95: | ||
== Исходный код == | == Исходный код == | ||
| - | Скачать код MATLAB можно здесь: [https://mlalgorithms.svn.sourceforge.net/svnroot/mlalgorithms/ | + | Скачать код MATLAB можно здесь: [https://mlalgorithms.svn.sourceforge.net/svnroot/mlalgorithms/Group774/Savinov2010Genetic/]. <br /> |
| - | + | Скачать реальные данные по опционам можно здесь: [https://mlalgorithms.svn.sourceforge.net/svnroot/mlalgorithms/Group774/Savinov2010Genetic/realOptions2D.txt realOptions2D.txt]. | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | Скачать реальные данные по опционам можно здесь: [https://mlalgorithms.svn.sourceforge.net/svnroot/mlalgorithms/Savinov2010Genetic/realOptions2D.txt realOptions2D.txt]. | + | |
== Смотрите также == | == Смотрите также == | ||
Текущая версия
|
Генетический алгоритм — эвристический алгоритм поиска, используется для решения задач оптимизации. Название обусловлено смыслом эвристики: моделируется биологический естественный отбор, степень приспособленности определяется значением целевой функции. Возможно применение генетического алгоритма для выбора признаков линейной регрессии, поскольку данная задача допускает оптимизационную формулировку (если множество порождающих функций зафиксировано).
Постановка задачи
Задана выборка — множество значений свободных
переменных и множество
соответствующих им значений зависимой переменной.
Задано множество порождающих функций линейной регрессионной модели
где
. Введем обозначения:
при
Требуется решить следующую задачу:
Описание алгоритма
Свободные параметры: размер популяции, доля кроссинговера (доля особей одного поколения, подвергающихся кроссинговеру).
- Инициализация (создание начальной популяции необходимого размера). Хромосомы особей представляют собой битовые векторы со случайными компонентами. Счетчик числа итераций:
Проверка условий завершения: либо
превысило максимально допустимое значение, либо изменения целевой функции SSE для лучшей особи в популяции за некоторое число итераций оказалось слишком малым.
- Селекция (выбор особей для следующего поколения). Селекция выполняется на основе оценки SSE всех особей. Используемый механизм таков: на единичном отрезке каждой особи выделяется участок, длина которого пропорциональна SSE. Далее алгоритм-селектор движется вдоль отрезка с фиксированным шагом, выбирая каждый раз ту особь, над которой находится. Начальная точка выбирается случайно, при этом начальное расстояние до левого конца должно быть меньше шага.
- Кроссинговер. Выбирается число особей, равное доли кроссинговера * размер популяции. С парами особей выполняется так называемый кроссинговер с маской (см. Оператор скрещивания). В результате этой операции каждая аллель (т. е. позиция в хромосоме особи) случайным образом заполняется геном 1-ого или 2-ого родителя.
- Мутация. Для каждой особи в каждой аллели с вероятностью 0.01 происходит мутация, которая выражается в замене текущего гена на 0 или 1 случайным образом (равновероятно).
- Возвращение на шаг 2.
Замечание
Существует еще одна модификация оператора кроссинговера - одноточечная, когда случайным образом выбирается позиция в хромосоме, и хромосомы особей обмениваются подстроками, заключенными между этой позицией и правым концом строки (см. Оператор скрещивания). Для этой модификации справедлива теорема схемы, которая имеет наглядную интерпретацию в терминах рассматриваемой задачи. Кратко изложим основную идею.
Пусть имеется какая-то начальная популяция. Рассмотрим некоторую совокупность признаков, не очень большую и имеющую не очень большое расстояние между крайними левыми и правыми позициями в хромосоме, дающую приближение в смысле SSE лучше, чем в среднем по данной популяции. Тогда количество появлений такой совокупности признаков в последующих популяциях будет расти экспоненциально на протяжении некоторого числа популяций. Таким образом, наборы признаков с высоким качеством сохраняются и распространяются по популяции.
Однако в данном исследовании используется кроссинговер с маской, для которого теорема схемы формально не может применяться.
Вычислительный эксперимент
Показана работа алгоритма на реальных и модельных данных.
Модельные данные
Данные порождались следующим образом (D - дисперсия):
x = [ 1:0.5:20 ]'; y = x + x.^2 + sin( x ) + log( x ) + x.*log( x ) + ( x.^2 ).*sin( x ) + sqrt( D ) * randn( size( x, 1 ), 1 );
Выборка делилась на training и testing части в соотношении 60%:40% случайным образом. Исследовались 2 эффекта: ложная (ранняя) сходимость и переобучение.
Результаты на модельных данных
Использовался следующий набор порождающих функций (всего 12):
В наборе не было следующих функций, участвовавших в порождении:
Случай небольшой дисперсии (D=1)
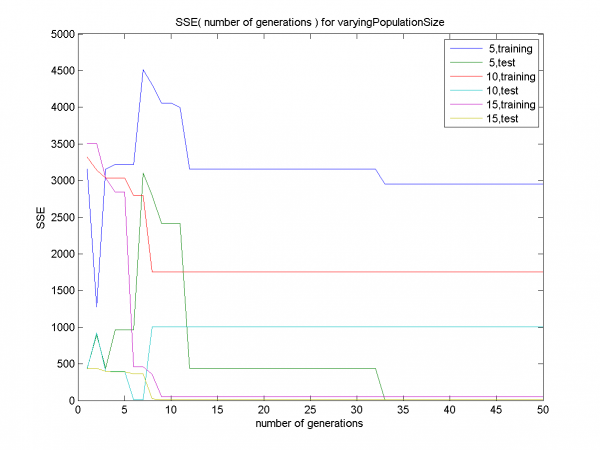
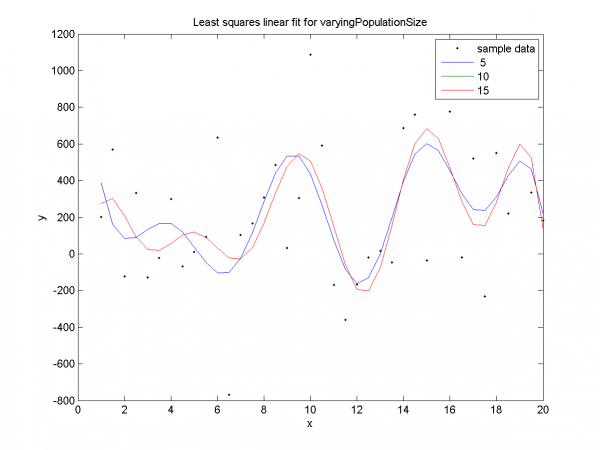
График для размера популяции 5 демонстрирует раннюю сходимость на обучающем множестве (training,5). Графики для размера популяции 10 демонстрируют раннюю сходимость и переобучение (т.е. SSE мало на обучающем множестве и велико на контрольном). Графики для размера популяции 15 показывают сходимость без переобучения.
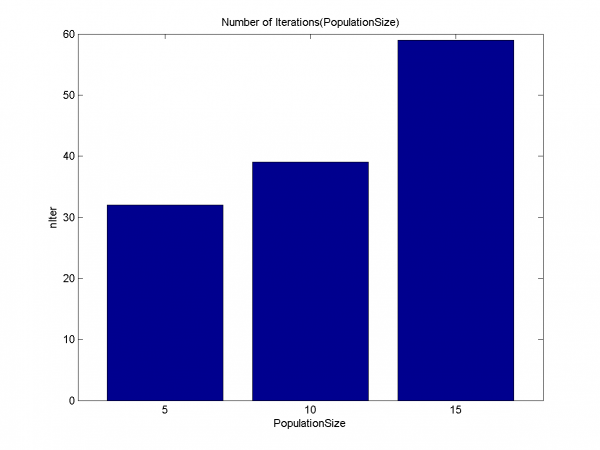
Подтверждение ранней сходимости для размеров популяции 5, 10. Дольше всего алгоритм работает для значения 15.
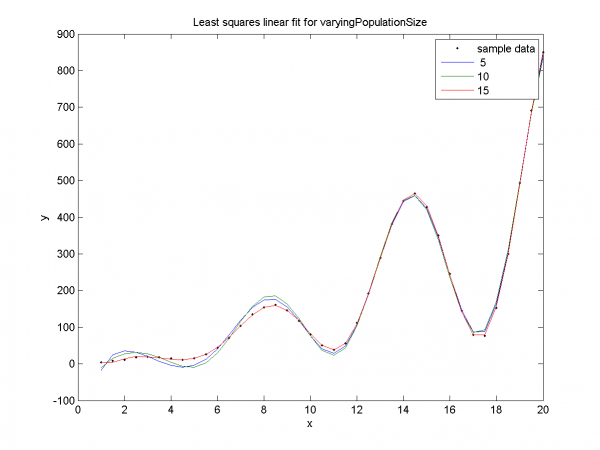
Наглядная демонстрация ранней сходимости и переобучения. Для размеров популяции 5, 10 SSE мало на training-части и велико на test-части. Для размера популяции 15 - подтверждается отсутствие ранней сходимости, переобучения.
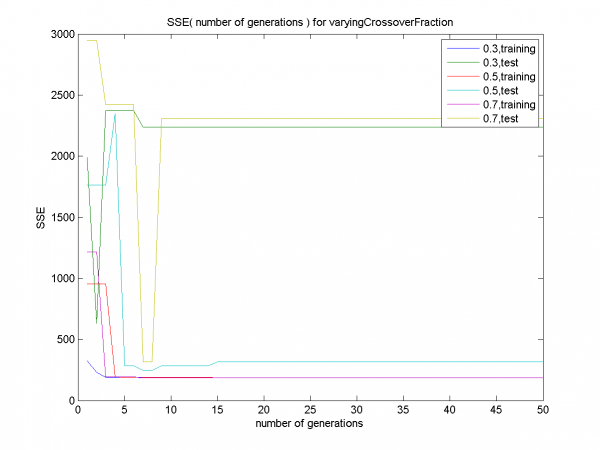
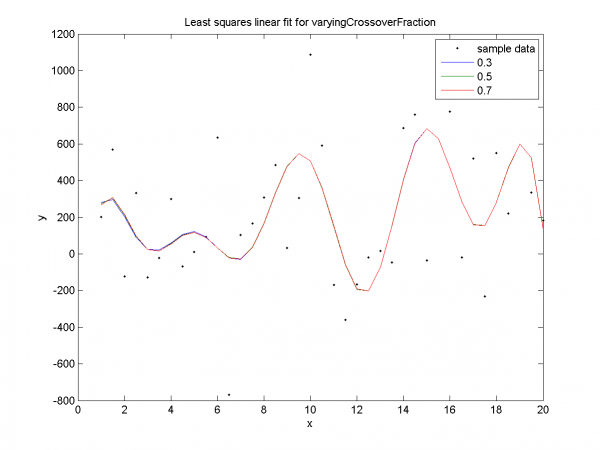
Графики для долей кроссинговера 0.3, 0.7 характеризуются переобучением. Для доли кроссинговера 0.5 переобучение не наблюдается, только ранняя сходимость.
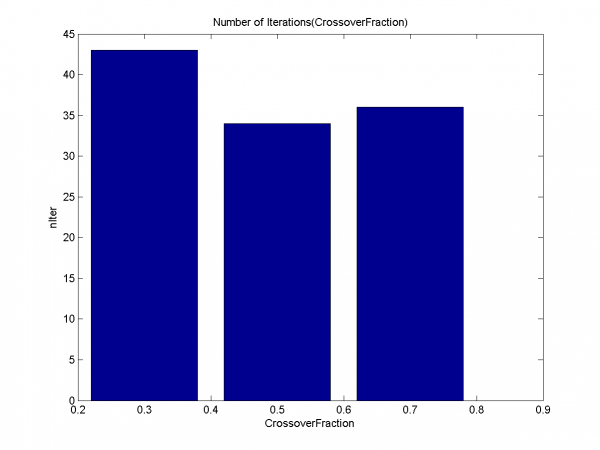
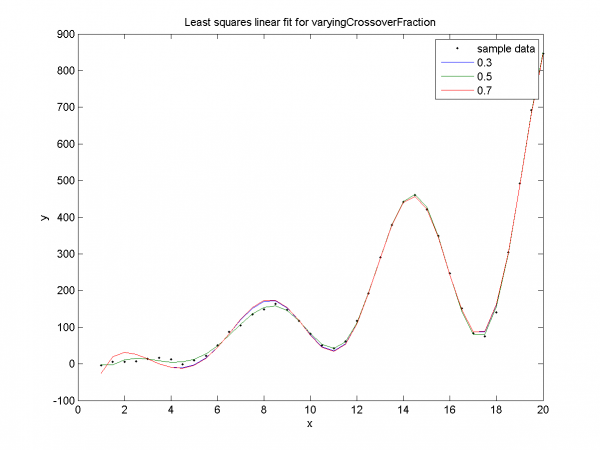
Наглядная демонстрация переобучения для долей кроссинговера 0.3, 0.7. Для значения 0.5 нет явного переобучения, как уже было замечено ранее.
Выводы
Чем больше размер популяции, тем менее вероятно переобучение и ранняя сходимость. Оптимальное значение доли кроссинговера равно 0.5.
Случай значительной дисперсии (D=160000)
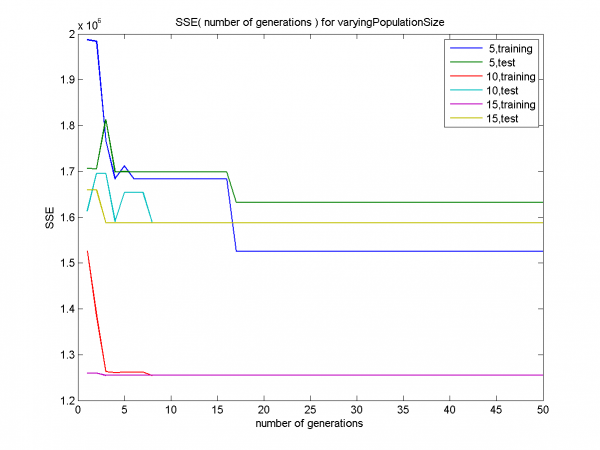
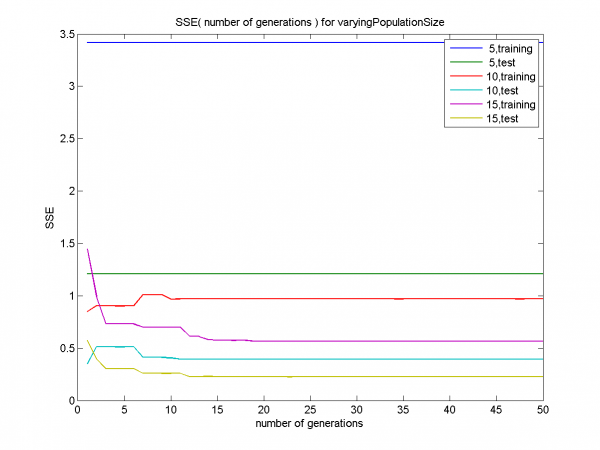
Видно, что для размера популяции 5 результатом работы алгоритма является точка пространства поиска с SSE значительно большим, чем для размеров популяции 10, 15.
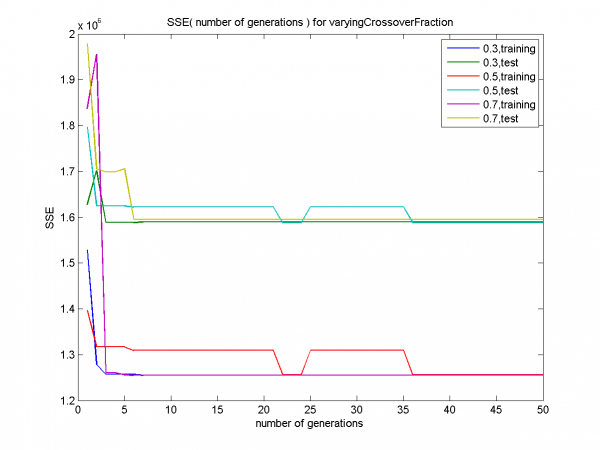
Начиная с 36-ой итерации все графики сливаются.
Четко видно отклонение синей линии, соответствующей размеру популяции 5, от линий, соответствующих значениям 10, 15 (эти линии сливаются). Графики SSE для всех значений, приведенные ранее, показали, что SSE для итоговой точки при значении 5 больше SSE в случаях 10, 15.
Все 3 графика сливаются, как уже было замечено ранее.
Выводы
При значительном шуме зависимость эффективности от доли кроссинговера очень слабая. Однако зависимость от размера популяции по-прежнему заметна, чем больше значение этого параметра - тем лучше.
Реальные данные
Выборка с улыбкой волатильности. 2 независимые переменные: время и страйк, зависимая переменная - волатильность. Выборка делилась как и прежде.
Результаты на реальных данных
Использовался следующий набор порождающих функций (всего 26):
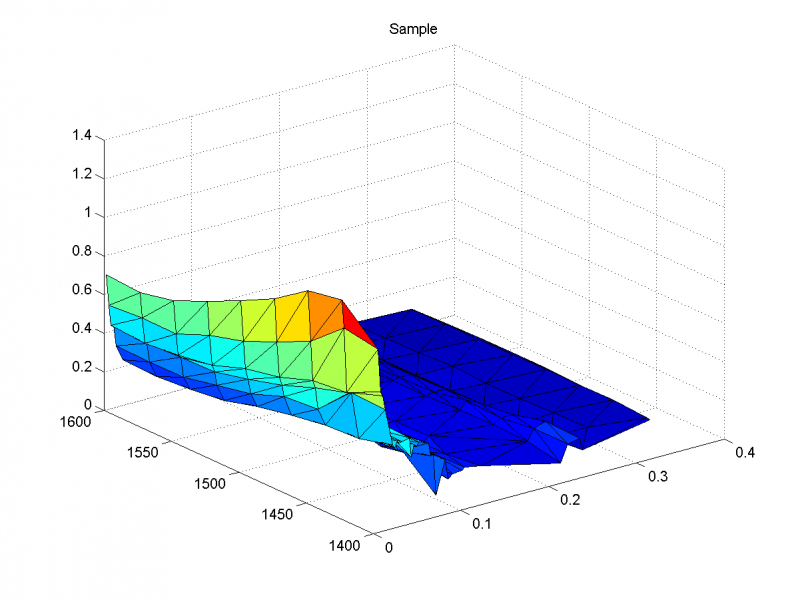
Результат с наименьшим SSE - при размере популяции 15, с наибольшим SSE - при размере популяции 5.
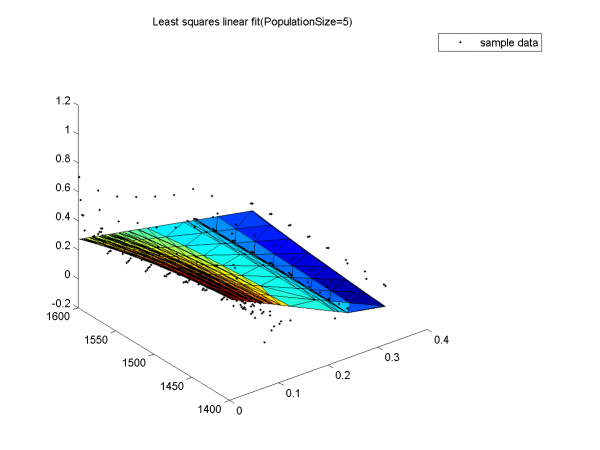
Результат с наибольшим SSE. Никакого сходства с данными.
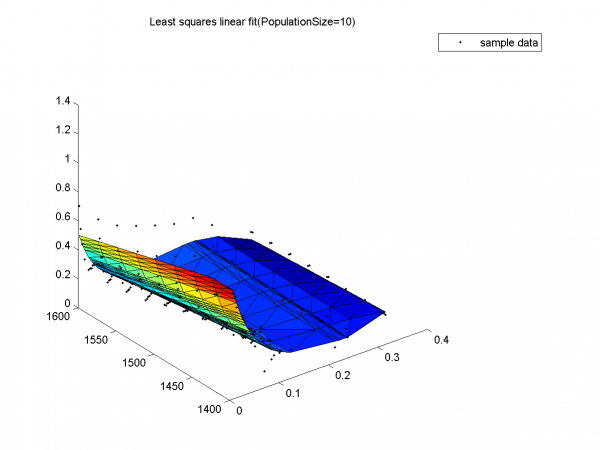
Неплохой результат, но видно, что красный край недостаточно загнут вверх относительно исходных данных, что выражается в большом значении SSE по сравнению с результатом для размера популяции 15.
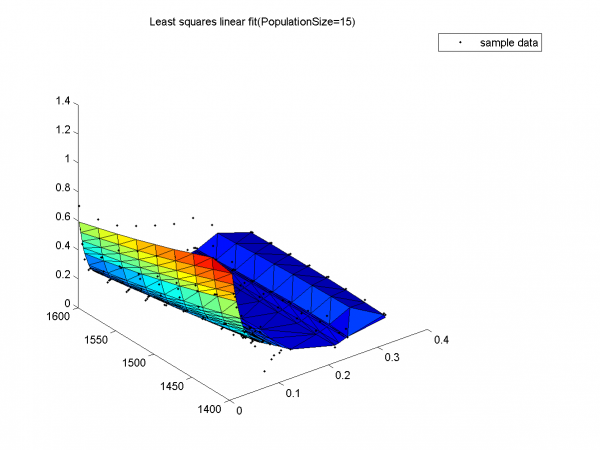
Результат с наименьшим SSE.
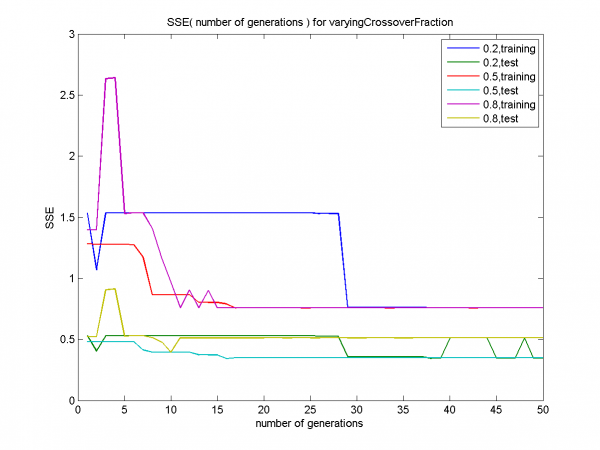
Разницы в эффективности для различных значений доли кроссинговера нет. Для всех 3 значений training-графики быстро сливаются (начиная с 30-ой итерации), test-графики достаточно близки.
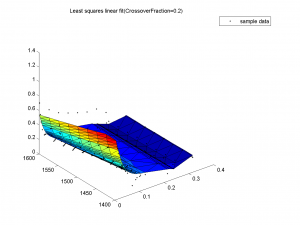
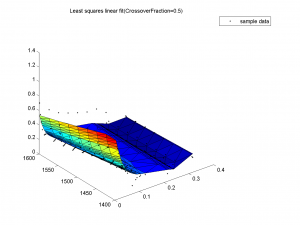
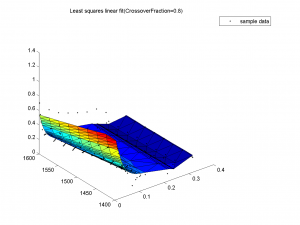
На всех 3 графиках видно, что поверхности почти не отличаются, все очень похожи на данные выборки.
Выводы
Подтверждаются выводы, сделанные для модельных данных с большой дисперсией.
Исходный код
Скачать код MATLAB можно здесь: [1].
Скачать реальные данные по опционам можно здесь: realOptions2D.txt.
Смотрите также
- Генетический алгоритм
- Теорема схемы
- Регрессионный анализ
- Линейная регрессия (пример)
- Метод наименьших квадратов
Литература
- Zbigniew Michalevicz, "Genetic Algorithms + Data Structures = Evolution Programs" (chapter 3: "GAs: Why Do They Work?").
- Melanie Mitchell, "An Introduction to Genetic Algorithms".
| | Данная статья была создана в рамках учебного задания.
См. также методические указания по использованию Ресурса MachineLearning.ru в учебном процессе. |

